研究人员预测原子力显微镜基底上的蛋白质位置
金泽大学的研究人员在《分子生物科学前沿》中报告了一种基于静电相互作用预测蛋白质在 AFM 基底上的位置的计算方法
使用原子力显微镜(AFM)观察生物分子结构,以及在高速 AFM (HS-AFM) 实验中直接可视化功能构象动力学,极大地促进了对纳米尺度生物过程的理解。
在实验中,生物样品沉积在支撑表面(AFM基底)上,并通过探针扫描来检测分子形状及其动态变化。在 HS-AFM 下观察蛋白质动力学是在将结构固定在支撑表面上与同时防止固定引起的过强扰动之间的微妙平衡。
在每次 AFM 观察开始时,将生物分子样品放置在支撑表面上并控制其正确附着的过程是一个挑战。通过缓冲液的化学成分,可以改变样品和底物之间的相互作用。这种表面修饰通常对于 AFM 成功观察蛋白质结构及其功能运动至关重要。然而,样品的分子取向是“先验”未知的,并且由于图像空间分辨率的限制,很难从“后验”分析中推断出来。
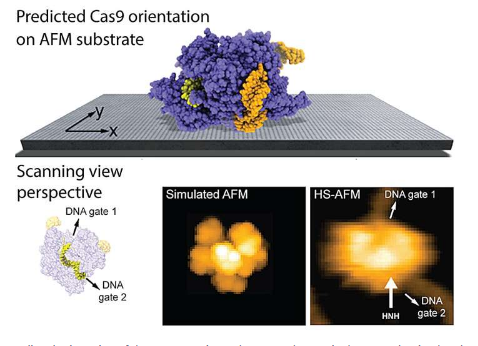
金泽大学的 Romain Amyot、Noriyuki Kodera 和 Holger Flechsig 现在开发了一种物理模型,可以根据静电相互作用预测生物分子结构在 AFM 基底上的位置。该方法考虑了 AFM 实验中常用的基材(云母、APTES-云母、脂质双层)并考虑了缓冲条件。
在计算机模拟中,对 AFM 基底上大量可能的分子排列进行采样,并通过评估相应的相互作用能量,确定最有利的排列。此外,该分析可以预测尖端扫描下的成像稳定性。
作者提供了新方法的几种应用,并获得了模型预测与之前蛋白质 HS-AFM 成像实验的显着一致性。例如,这些发现可以解释为什么 HS-AFM 对 Cas9 核酸内切酶(一种在基因工程应用中发挥关键作用的蛋白质)的观察可以可靠地可视化目标 DNA 和 Cas9 的功能性相对运动,并在单分子水平捕获 DNA 切割事件。
此外,正如 ATP 驱动的伴侣机 ClpB 所证明的那样,该模型可以解释缓冲条件如何影响样品-基质复合物的稳定性,并验证之前 HS-AFM 实验的观察结果。
总之,新方法甚至可以在实际实验之前利用大量可用的生物分子结构数据来预测 AFM 基板上的样品放置情况,并且还可以应用于 AFM 成像数据的实验后分析。
所开发的方法在免费提供的 BioAFMviewer 软件包中实施,为广泛的 BioAFM 社区的应用提供了便利的平台。
