生成式人工智能加速抗病毒药物开发和药物发现的潜力
在一项新研究中,来自IBM、牛津大学和DiamondLightSource的研究人员表明,IBM的AI模型MoLFormer可以针对包括SARS-CoV-2在内的多种目标病毒蛋白生成抗病毒分子,从而可以加速药物发现过程并支持我们的研究。应对未来的流行病。
研究结果发表在《科学进展》杂志上的一篇新论文中,在论文提交时,牛津大学研究人员成功验证了11种分子的抗病毒特性。这一突破有可能在下一次危机中更快地为人们提供药物,并为紧急、危及生命的疾病提供治疗方法。
在大流行初期,IBM的一组计算机科学家希望探索是否可以使用生成式AI来设计前所未见的分子来阻止SARS-CoV-2(这种导致COVID-19的病毒)。DavidStuart,牛津大学临床医学系结构生物学系主任,英国国家同步加速器DiamondLightSource生命科学主任,是HIV、SARS和埃博拉等病毒病原体方面的权威解释说他最初对此表示怀疑。他说:“通过人工智能,可以获取蛋白质序列,凭空提取出与病毒3D位点结合的化学物质,这种想法似乎不太可能实现。”
然而,他和结构生物学专家、Diamond生命科学副主任MartinWalsh加入了IBM团队,在三年的时间里证明了生成式AI可以“凭空为抗病毒药物寻找可行的起点,”与乌克兰化学品供应商EnamineLtd.以及牛津大学的其他研究人员合作。
由于生成模型也是一个基础模型,经过大量原始数据的预先训练,因此它具有足够的通用性,可以为多个蛋白质靶点创建新的抑制剂,而无需额外的训练或对其3D结构的任何了解。
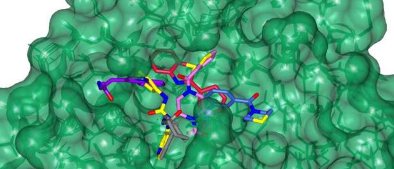
Stuart和Walsh小组已开始研究两种重要的SARS-CoV-2蛋白,即刺突蛋白和主要蛋白酶。利用这些目标,该团队在很短的时间内找到了四种潜在的COVID-19抗病毒药物。然后,这项工作利用Diamond的高通量大分子晶体学光束线来可视化人工智能生成的化合物的子集如何与主要蛋白酶结合。
他们的工作在ScienceAdvances上的新论文中得到了展示,IBM还发布了一个基于Web的界面,用于与IBMCloud中的模型和化学基础模型进行交互。
该团队表示,经过验证的分子在公司将其转化为药物之前还需要克服更多障碍,包括临床试验。但即使人工智能产生的“热门产品”从未转化为实际药物,这项工作也证实了生成式人工智能在未来的药物开发中可以发挥重要作用,尤其是在危机时期。
该研究的共同高级作者、IBM研究院研究员PayelDas表示:“开发和验证这些方法需要时间,但现在我们有了工作流程,我们可以更快地生成结果。”“当下一种病毒出现时,生成人工智能可能在寻找新疗法中发挥关键作用。”
Diamond的联合资深作者MartinWalsh表示:“生成与感兴趣的药物靶标具有高亲和力结合的初始化合物,可以加速基于结构的药物发现流程,并支持我们为未来的流行病做好更好准备的努力。”
研究人员在称为变分自动编码器(VAE)的生成式人工智能架构上构建了他们的模型“分子受控生成”(或CogMol)。VAE将原始数据编码为压缩表示,然后将其解码或转换回原始样本的统计变化。他们的模型是在以文本字符串表示的大型分子数据集以及有关蛋白质及其结合特性的一般信息上进行训练的。但他们故意遗漏了有关SARS-CoV-23D结构或已知与其结合的分子的信息。他们的目标是为他们的生成基础模型提供广泛的知识基础,以便可以更轻松地将其部署到以前从未见过的分子设计任务中。
他们的目标是找到能够与两个新冠病毒蛋白靶点结合的类药物分子:将病毒传播到宿主细胞的刺突,以及有助于病毒传播的主要蛋白酶。尽管当时已经发现了这两种蛋白质的3D结构,但IBM研究人员选择仅使用源自DNA的氨基酸序列。通过以这种方式限制自己,他们希望模型能够在不知道目标形状的情况下学会生成分子。
研究人员仅将每个蛋白质靶标的氨基酸序列输入CogMol,三天内生成了875,000个候选分子。为了缩小范围,研究人员通过逆合成平台IBMRXNforChemistry运行候选者,以了解合成这些化合物需要哪些成分。根据平台预测的配方,他们为每个目标选择了100个分子。Enamine的化学家进一步将每个目标的列表削减为四个分子,选择那些被认为最容易制造的分子。
合成了八种新型分子后,Enamine将它们运往牛津大学,在ChrisSchofield教授和教授GavinScreaton的实验室中测试它们破坏两种蛋白质靶标功能的能力。。钻石产生的强X射线束比太阳亮100亿倍,用于可视化化合物如何与蛋白质相互作用以使其功能失活。这些新型化合物在靶点抑制和活病毒中和试验中进行了进一步测试。两种经过验证的抗病毒药物以主要蛋白酶为目标;另外两种不仅针对刺突蛋白,而且被证明能够中和所有六种主要的新冠病毒变体。“你会得到一张地图,准确地显示事物结合的位置,然后砰!你就得到了确认,”斯图尔特说。
CogMol是IBM此后开发的几个化学基础模型之一。最大的MoLFormer-XL接受了超过11亿个分子的数据库的训练,目前被Moderna用于设计mRNA药物。该研究的共同高级作者、IBM研究院研究员、牛津大学教授杰森·克雷恩(JasonCrain)表示:“我们使用生成基础模型为加速抗病毒药物的开发创造了有效的起点,而该模型对其蛋白质靶标知之甚少。”“我希望这些方法将使我们能够在未来更快、更便宜地制造抗病毒药物和其他急需的化合物。”
尽管研究人员的重点是验证新冠病毒的抗病毒药物,但他们认为这些方法可以扩展到继续变异的现有病毒,如流感或尚未出现的病毒。“如果你想为下一次大流行做好准备,你需要作用于蛋白质不同位点的药物,”斯图尔特总结道。“病毒逃脱变得更加困难。”
